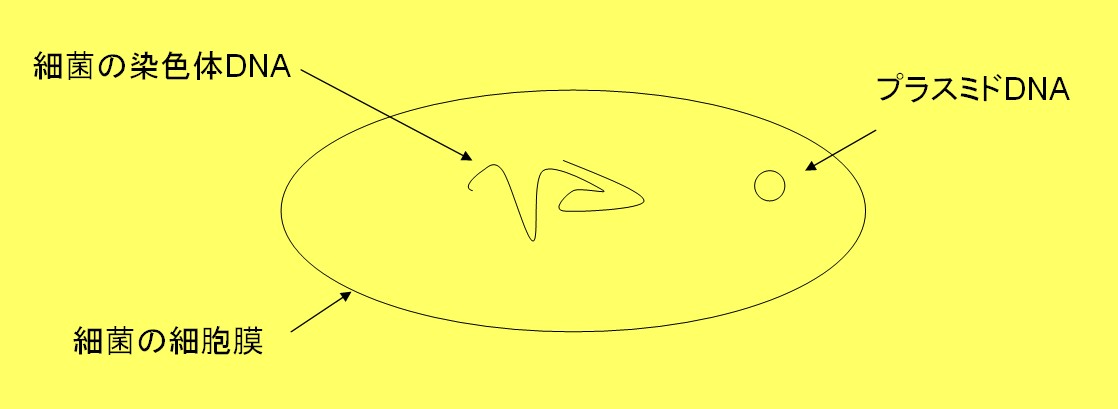

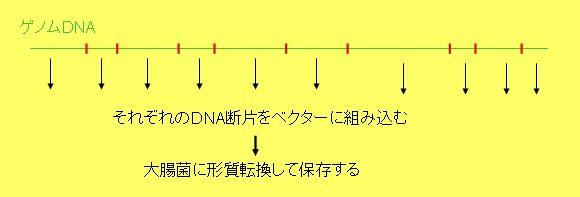
プラスミドDNAの1箇所を切断し、そこにDNA断片を組み込む。 できあがった人工組換えDNAもプラスミドとして作用するので、 大腸菌の中でプラスミドを増やすことによって、DNA断片を増幅できる。 これをDNAクローニングと呼ぶ。
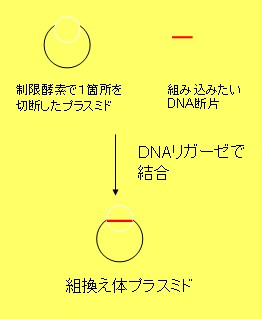
DNAをある特定の塩基配列で切断するのが制限酵素である。 切断されたDNAどうしを貼りつける酵素がDNAリガーゼであり、 貼りつける反応をライゲーションと呼ぶ。

ベクターとして使用するプラスミドに抗生物質耐性遺伝子を組み込んでおくと、 そのプラスミドを持つ大腸菌が抗生物質に耐性になる。 従って、プレート培地や液体培地にその抗生物質を入れておいてやると、 プラスミドを持つ大腸菌のみを選択的に増幅することができる。
抗生物質としてはアンピシリン、テトラサイクリン、クロラムフェニコールなどが使用される。 大腸菌は体内のプラスミドを体外に排除する作用があるので、 プラスミドを持つ大腸菌を保持するために、必ず培地に必要な抗生物質を加えるようにする。
増幅したいDNAとベクターをライゲーションする時に、 ベクターが自分自身とライゲーションするセルフライゲーションが起きることが多い。 セルフライゲーションしたベクターを選別するためにブルーホワイトセレクションが考案された。
ベクターの増幅したいDNAを組み込む点を含む両側に、lacZ遺伝子を配置しておく。 lacZ遺伝子はIPTGとX-galの存在下でlacZタンパク質を発現し、大腸菌コロニーを青くする。
lacZ遺伝子の中に増幅したいDNAがライゲーションされると、転写が抑制されたり、 正常なlacZタンパク質が発現しないために、大腸菌コロニーは白いままである。
そこで、プレート培地にIPTGとX-galを添加して、形質転換された大腸菌をまくと、 DNAが組み込まれたプラスミドを持つ大腸菌は白いコロニーになり、 セルフライゲーションしたプラスミドを持つ大腸菌は青いコロニーとなる。
ただし、培養時間が短かったりすると青色がきれいに発現しなかったり、 短いDNA断片をクローニングしたベクターを持つ大腸菌では、うすい青色を呈する場合もある。
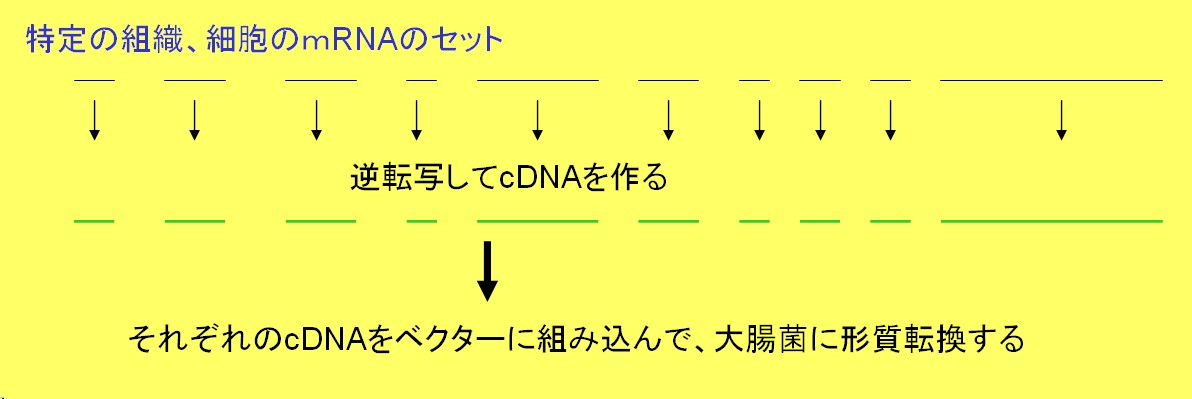
mRNAに逆転写酵素を加えることによって1本鎖のDNAを作ることができる。 これを1本鎖のcDNAと呼ぶ。 これにDNAポリメラーゼを作用させれば2本鎖のcDNAが作成できる。 このcDNAをプラスミドやラムダファージに組み込んでライブラリと したものがcDNAライブラリである。
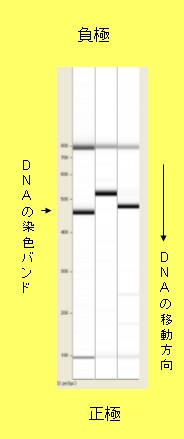

適当な長さの塩基配列を持ったオリゴヌクレオチドをRIでラベルするか、 蛍光色素や化学発光物質などで標識をする。これをプローブと呼ぶ。 プローブが相補的なライブラリのDNAに結合するように反応させると、 RIあるいは蛍光シグナルにより、ライブラリのどのクローンが探し出したい DNA断片を持っているかがわかる。 このようにライブラリから必要なDNA断片を探し出すことをライブラリの スクリーニングと呼ぶ。
以上のような手法をコロニーハイブリダイゼーションと呼ぶが、そのほかにもハイブリダイゼーションを用いた手法がいくつかある。 ドットブロットハイブリダイゼーションはサンプルのDNAをナイロンなどでできたメンブレンに転写、固定し、プローブをハイブリさせる方法で、 おもに特定のDNA配列の有無の検査に使用される。
サザンハイブリダイゼーションはサンプルをアガロース電気泳働し、そのゲル上のDNAをメンブレンに転写、固定し、プローブをハイブリさせる方法で、 おもに特定のDNA配列を含むDNA断片の長さと濃度を調べるのに使用される。ノーザンハイブリダイゼーションはそのRNA版である。
DNAマイクロアレイはスライドグラスに多数の遺伝子のcDNAをスポットしておき、それに対して各組織や細胞から作成したcDNAをハイブリさせる方法で、 ガン組織などの特定の組織や細胞に特異的に発現している遺伝子を検索するのに用いられる。

PCR法はDNAクローニング法よりも短時間で効率的に特定のDNA断片を増幅することができる。 また、DNA断片に対する特異性も高い。 しかし、プライマーを作成するための情報が必要であり、 プライマーの設計が悪いと、非特異的増幅が起きたり、 プライマーダマーが形成されることがある。
PCRによる増幅は指数関数的であり、PCR後の産物の濃度は一般には初期鋳型量に比例しない。 しかし、特殊なリアルタイムPCR法を用いることによって、初期鋳型量を定量することが可能であり、mRNAの定量に広く使用されている。
ハイブリダイゼーションの技術を用いてマッピングした地図を染色体物理地図と呼ぶ。
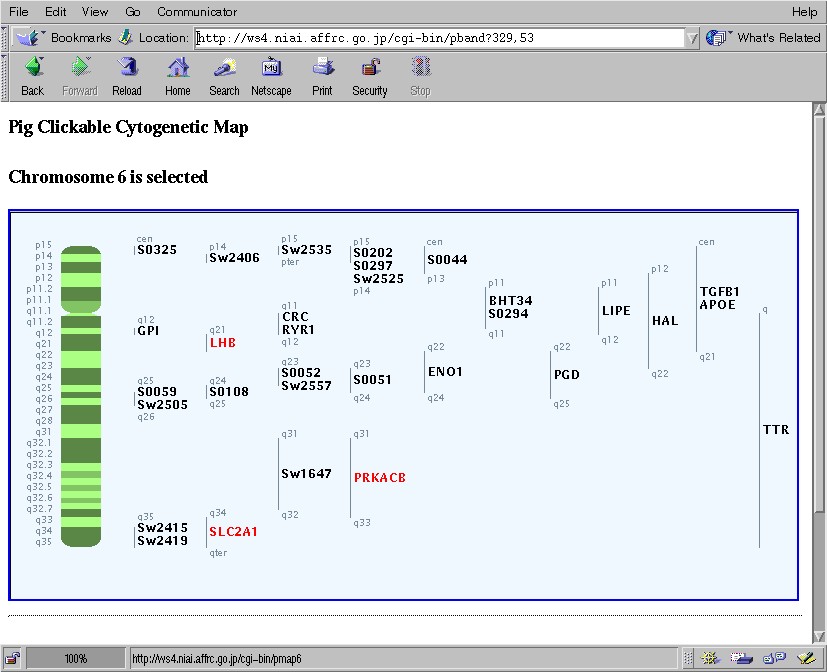
連鎖と組換え(次次回で説明する)の理論に基づいて、 ゲル電気泳動の手法などを使ってマッピングした地図を連鎖地図という。
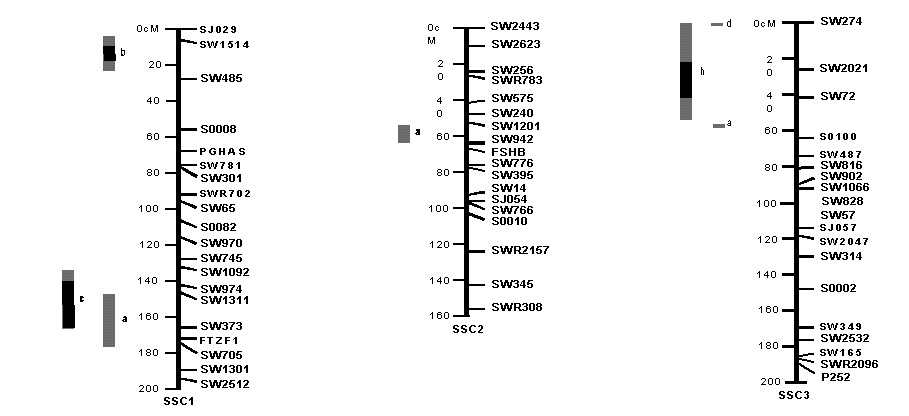
さらに、ゲノムライブラリの個々のクローンをもとのゲノムDNAを復元するように並べたものをコンティグマップと呼ぶ。
多数のDNAマーカーと遺伝病との連鎖関係の情報から、遺伝病の責任遺伝子のゲノム上の位置がわかる。 さらに、乳量や肉質といった経済形質とDNAマーカーとの連鎖関係から、経済形質に関与する遺伝子のゲノム上の位置を明らかにすることが試みられている。
500bp程度読んで、その情報から新しいプライマーを設定してその先を読むという作業を順次行う方法をprimer walkingと呼ぶ。 この場合、正確を期すには両側のDNA鎖を読む必要がある。
ゲノムのフルシーケンスを決める場合などには、ショットガンという手法を用いる。 これはクローン全体を超音波処理して400ー500bp程度に断片化し、それぞれの断片をプラスミドのサブクローニングする。 そして、そのショットガンライブラリのプラスミドに挿入されたDNA断片の塩基配列を片端から読んでいく。 もとのクローンの全長の3-4倍程度の配列を読んで、 重複している部分を頼りにコンピュータを利用してもとの配列を再現していく。 primer walkingと異なって、機械化できる部分が多いので、大規模なシーケンスにはショットガンの方がコストパフォーマンスに優れている。

最終更新年月日 2009年7月21日