
佐賀大学農学部応用生物科学科 生物情報処理演習 講義テキスト 科目ホームページ
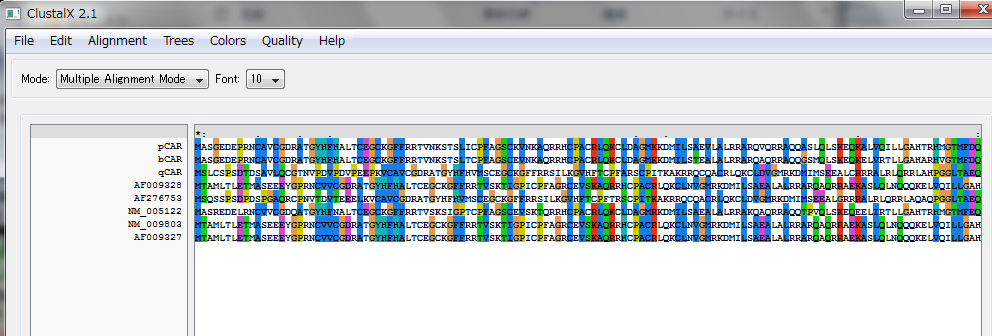 Alighnment/Do Complete Alighnmentを選択すると、マルティプルアライメント作成用のガイドツリーのファイルと作成されたアライメントファイルのフォルダーとファイル名が指定できます。
OKをクリックするとマルティプルアライメントが実行されます。
Alighnment/Do Complete Alighnmentを選択すると、マルティプルアライメント作成用のガイドツリーのファイルと作成されたアライメントファイルのフォルダーとファイル名が指定できます。
OKをクリックするとマルティプルアライメントが実行されます。
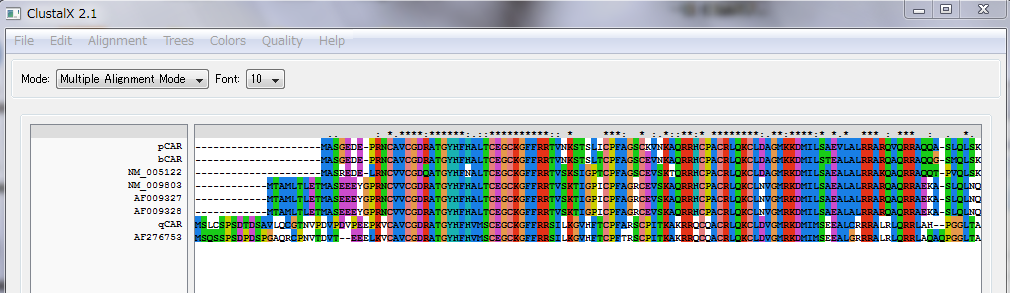 このように画面上ではアミノ酸ごとに色分けされて見やすくなっています。
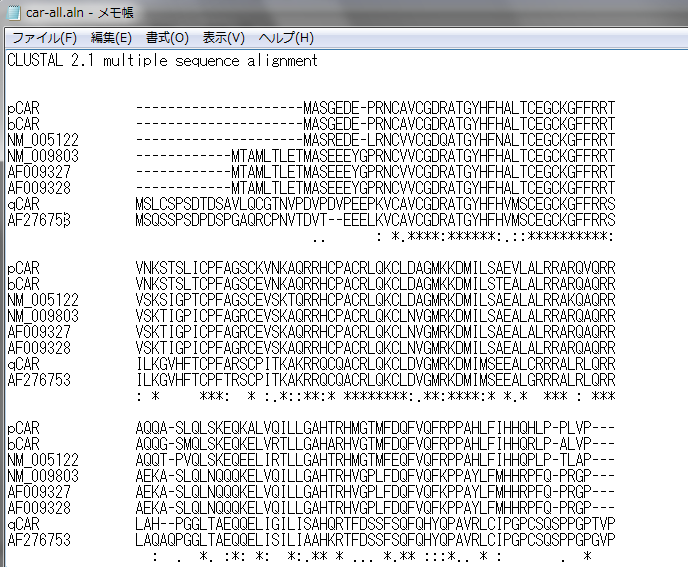
作成されたアライメントはalnという拡張子を持つテキストファイルとして出力されます。
このように画面上ではアミノ酸ごとに色分けされて見やすくなっています。
作成されたアライメントはalnという拡張子を持つテキストファイルとして出力されます。
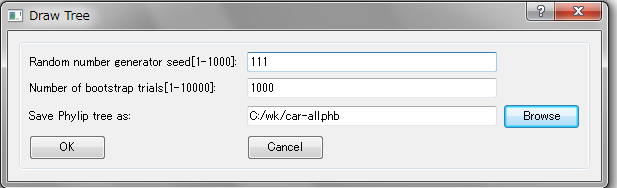 乱数の種(seed)が同じであればいつも同じ結果になりますが、種が違うと微妙に違う結果になることもあります。
ここではブートストラップ1000回の系統樹を作成しています。
乱数の種(seed)が同じであればいつも同じ結果になりますが、種が違うと微妙に違う結果になることもあります。
ここではブートストラップ1000回の系統樹を作成しています。
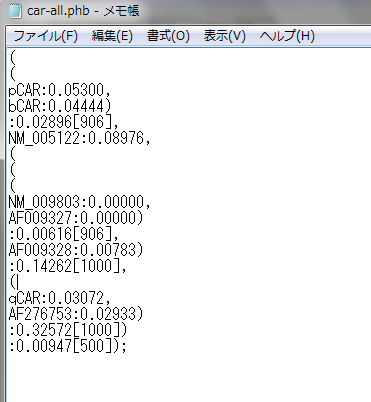 Clustal Xでは分子系統樹を描画する機能はなくphylip形式のテキストファイル(拡張子 phb)として出力されます。
phbファイルを描画するにはNJplotを用います。
Clustal Xでは分子系統樹を描画する機能はなくphylip形式のテキストファイル(拡張子 phb)として出力されます。
phbファイルを描画するにはNJplotを用います。
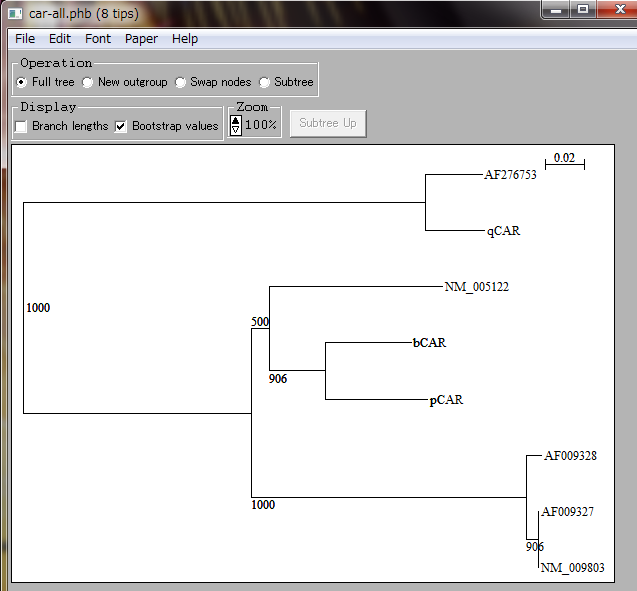 File/Open でphbファイルを読み込み、Bootstrap valuesにチェックを入れると、図のように系統樹のノードに数字が表示されます。
この数字はブートストラップの回数(ここでは1000回)のうち、この分岐になった回数を示しています。
この数字が大きいほど、このノードの分岐が確からしいということを表しています。
File/Open でphbファイルを読み込み、Bootstrap valuesにチェックを入れると、図のように系統樹のノードに数字が表示されます。
この数字はブートストラップの回数(ここでは1000回)のうち、この分岐になった回数を示しています。
この数字が大きいほど、このノードの分岐が確からしいということを表しています。

最終更新年月日 2019年1月7日