佐賀大学農学部応用生物科学科 生物情報処理演習 講義テキスト 科目ホームページ
そのためにはOpen Reading Frame(ORF)を確定する必要があります。
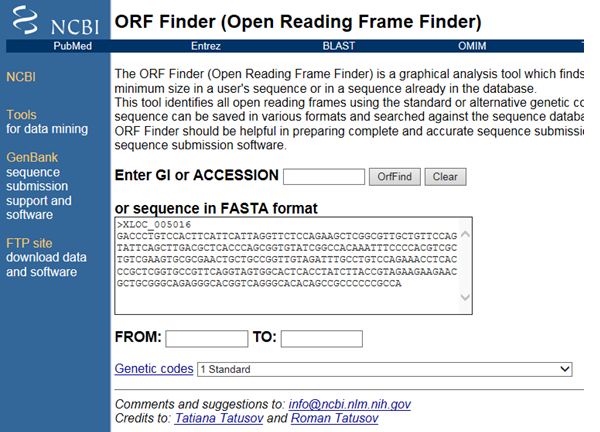
NCBIのORF Finderを利用すれば、プラス鎖とマイナス鎖両方について、1塩基ずつずらした3x2の計6通りで、可能性のあるアミノ酸配列をすべて表示してくれます。
通常は、そのなかで最長のアミノ酸配列を示すものが正しいORFになります。

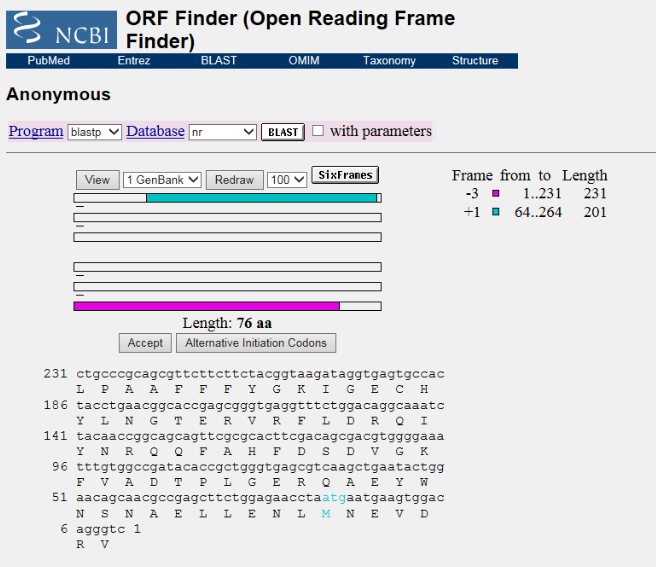
これはピンクのORFのアミノ酸配列を表示したものですが、開始コドンの位置がおかしいようです。
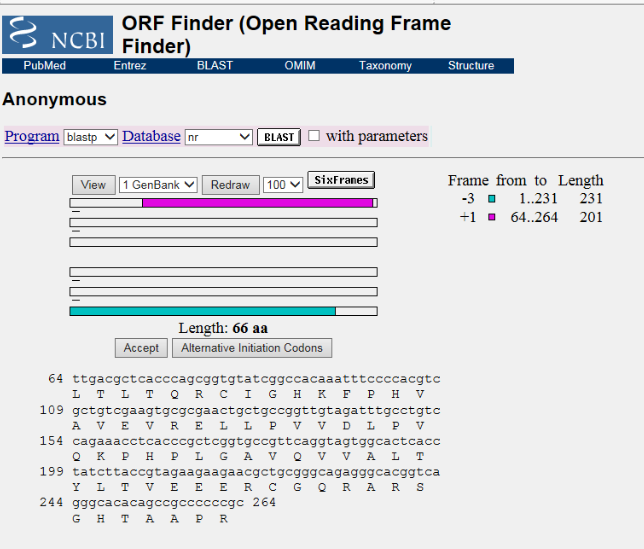
このORFには開始コドンが見当たりません。
実験において全長のmRNAが得られていない場合は5'部分が切断されて開始コドンが存在しない場合もありますが、利用するには注意が必要です。
しかし、短いORFしか表示されないものしかない場合は、そもそもこの遺伝子がタンパク質をコードしていないのかもしれません。
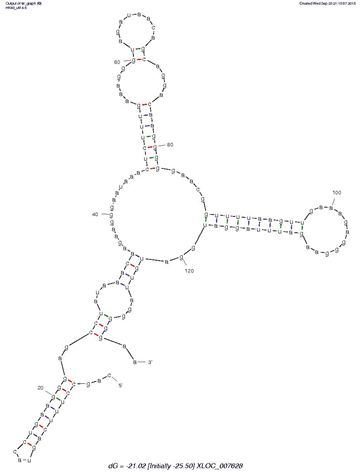
tRNAなどは特徴的な2次構造をしているので、mfoldなどを利用してRNAの2次構造を推定するといいでしょう。
また、RNA配列を塩基配列に対するblast検索にかけると、micro RNAなどにヒットすることもあります。
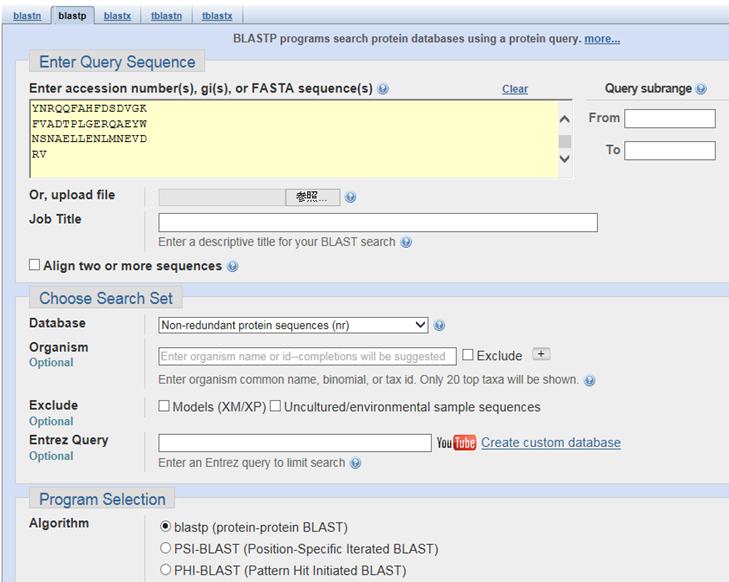
そのために、得られたアミノ酸配列をblast検索にかけます。
blast検索はNCBIのシステムが便利です。
既知の遺伝子であればblast検索でヒットするでしょう。
ただし類似度が低い場合や一致するのが配列の一部である場合は、別の遺伝子である可能性もあります。

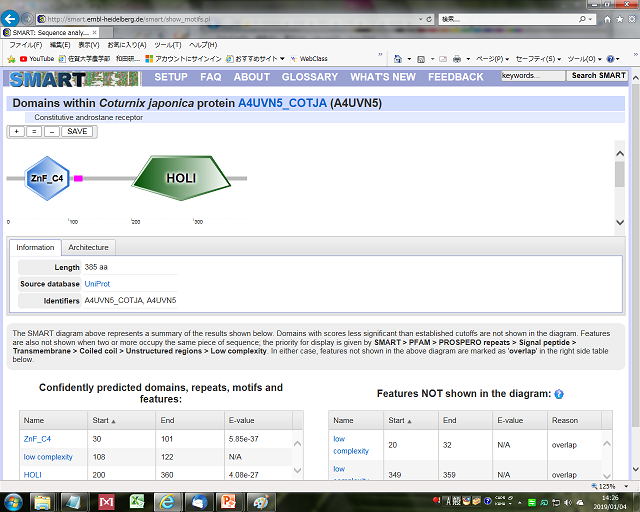
タンパク質にはモチーフやドメインといった多くの遺伝子で共通に現れるアミノ酸配列があり、これらの多くは機能が明らかになっています。
balst検索時にこれらのモチーフやドメインが表示される場合もありますが、SMARTなどの専用のソフトもあります。
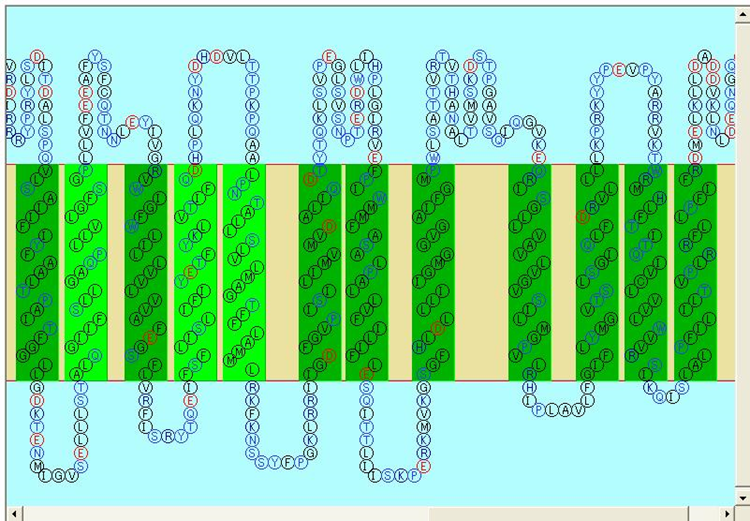
blastやSMARTなどでヒットしない場合はアミノ酸配列を物理化学的に解析してその機能を推測することも可能です。
1例としてSOSUIは疎水性のアミノ酸の連なりから膜貫通部位を推定するものです。

最終更新年月日 2019年1月4日